Par Céline Brochier
I. Introduction
1/ Quelques aspects historiques
.
Découverte
L’origine du terme « cellule » remonte au 17ième siècle. C’est le savant Robert Hooke qui fut à la fois physicien, chimiste, biologiste, astronome, inventeur, architecte, qui introduit le mot en biologie dans son ouvrage, les Micrographia (1667), qui décrit les observations qu’il a faites avec un microscope de sa fabrication. En particulier, en observant un morceau de liège, il remarqua des cavités délimitées par des parois de cellulose et de suber qu’il nomma « cellules ». Toutefois, ni Hooke, ni ses contemporains ne comprirent l’importance de cette notion et c’est seulement au XIXème siècle qu’elle s’imposa.
Théorie cellulaire
Le botaniste allemand Matthias Schleiden et son ami Theodor Schwann proposèrent, dans une note de 1839, l’hypothèse, preuves microscopiques à l’appui, que tous les êtres vivants, plantes, animaux, moisissures, sont constitués d’un ensemble d’unités de construction de même type, les cellules. La théorie cellulaire, devait donner naissance à une synthèse unificatrice qui constitue en biologie, au même titre que la théorie atomique en physico-chimie, une de ses plus solides fondations. La théorie cellulaire définit la cellule comme l’unité d’organisation structurale des systèmes vivants. Certains d’entre eux sont formés d’une unique cellule autonome (ce sont les organismes unicellulaires), tandis que d’autres sont formés de plusieurs cellules différenciées du point de vue de la forme et des fonctions (ce sont les organismes pluricellulaires). En 1855, à la suite des travaux de Robert Remak (1815-1865), Rudolf Virchow (1821-1902) formule le second axiome de la théorie cellulaire : toute cellule provient d’une autre cellule. On peut résumer la théorie cellulaire ainsi : tous les êtres vivants sont constitués d’unités microscopiques, les cellules et chaque cellule possède les propriétés caractéristiques du vivant.
2/ Chaque cellule possède les propriétés caractéristiques du vivant
Le problème de la définition de la cellule minimale est intimement lié à celle de la vie. Les recherches tournant autour de ce qui est vivant sont multiples : vie artificielle, vie-prébiotique, modèles cellulaires… Or paradoxalement, il n’existe pas de définition consensuelle de ce qui est vivant. Les définitions de ce qu’est la vie sont rares et ont souvent changé au cours du temps 1. Le problème majeur rencontré est que cette définition doit (1) être applicable largement (aux objets terrestres mais aussi à l’infinité des objets extra-terrestres), (2) être suffisamment précise pour permettre une distinction fine entre ce qui est vivant et ce qui ne l’est pas (des virus, une cellule morte, un cristal en croissance), et (3) ne pas être trop restrictive car elle doit inclure tout ce qui est vivant actuellement ainsi que les formes de vie hypothétiques antérieures. On pourra néanmoins retenir cette définition proposée par la NASA en 1997 est vivant « Un système qui est délimité sur le plan spatial par une membrane semi-perméable de sa propre fabrication, qui est capable de s’auto entretenir et de se reproduire en fabriquant ses constituants à partir d’énergie et/ou de matériaux bruts extérieurs. ». Toutes les cellules (considérées comme des systèmes vivants possèdent les trois caractéristiques du vivant : l’autoconservation (récupérer des éléments du milieu et les transformer en produits nécessaires à la survie), l’autorégulation (contrôler la fabrication de ces produits) et l’autoreproduction (produire un système ayant ces 3 caractéristiques).
3/ Caractéristiques des cellules actuelles
A) Stockage de l’information héréditaire sous forme d’ADN et sa transmission
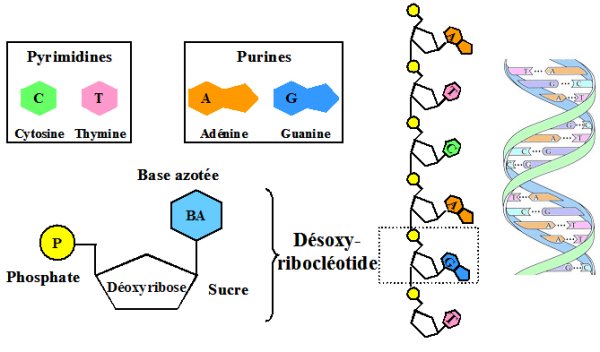
Toutes les cellules vivantes sans aucune exception connue conservent leur information génétique sous forme d’une ou plusieurs molécules d’ADN (acide désoxyribonucléique) double brin (Figure 1). Chaque brin est un polymère non ramifié de désoxyribonucléotides, eux-mêmes constitués d’un sucre (déoxyribose), relié à un groupement phosphate et à une base azotée (Figure 1). Il existe quatre types de bases donnant leur spécificité aux nucléotides : Adénine (A), Cytosine (C), Guanine (G) et Thymine (T). Les deux brins constituant la molécule d’ADN sont appariés par complémentarité des bases (Adénine/Thymine, Cytosine/Guanine).
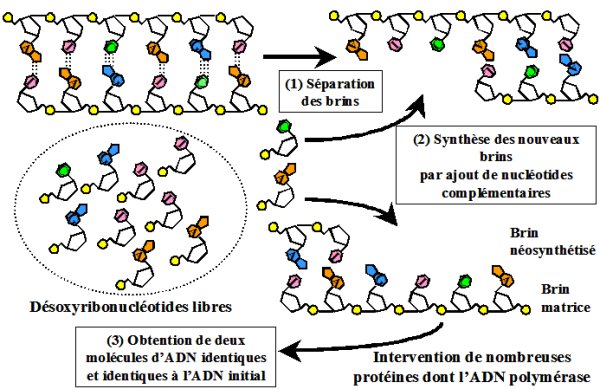
Lors de la reproduction cellulaire, processus par lequel une cellule mère va se diviser, l’intégralité de son information génétique est transmisse aux cellules filles qui portent donc la même information génétique que la cellule qui leur a donné naissance. Cela signifie que l’information génétique portée par la cellule mère doit être préalablement dupliquée. Ce processus duplication de l’ADN s’appelle la réplication. Chez toutes les cellules actuelles cette réplication se fait grâce à une matrice et à l’intervention de nombreuses protéines. Lors de la réplication les deux brins constituant la molécule d’ADN se séparent et chaque brin va servir de matrice aux nouveaux brins qui vont être synthétisés (Figure 2). L’ajout de nucléotides va se faire en suivant les règles d’appariement des bases (A avec T et C avec G). A la fin de la réplication, on obtient deux molécules d’ADN identiques et identiques à la molécule d’ADN initiale. On appelle ce mode de réplication de l’ADN semi-conservatif car chaque nouvelle molécule d’ADN est constituée d’un brin de la molécule d’ADN initial et d’un brin complémentaire néosynthétisé.
Figure 2 : Réplication de l’ADN
.
B) Transcription de l’information héréditaire sous la forme d’un intermédiaire l’ARN
L’ADN sert de support à l’information génétique (les gènes). Les gènes sont des segments d’ADN contenant les instructions nécessaires à la fabrication de deux types de molécules clés indispensables à toutes les cellules : l’ARN et les protéines. Cependant l’information contenue dans les gènes n’est pas utilisée directement par les cellules. Toutes les cellules actuelles recopient les parties de l’information génétique qu’elles souhaitent utiliser sous la même forme intermédiaire : l’ARN (acide ribonucléique). L’ARN est un polymère simple brin non ramifié de ribonucléotides, formés d’un sucre (le ribose), lié à un groupement phosphate et à une base azotée. La principale différence entre l’ADN et l’ARN (outre la nature du sucre qui est légèrement différente) tient à la nature des bases utilisées. Pour fabriquer de l’ARN les cellules utilisent quatre types de bases azotées : l’adénine (A), la cytosine (C), la guanine (G) et l’uracile (U). Contrairement à l’ADN on ne rencontre pas de thymine dans l’ARN mais de l’uracile. La fabrication des ARN se fait au cours de la transcription. La première étape est la séparation des brins d’ADN où est localisée l’information génétique à transcrire (Figure 3). Lors de la seconde étape le brin d’ARN est synthétisé à partir de la séquence d’ADN complémentaire contenant l’information à transcrire. Cette transcription fait appel à de nombreux acteurs protéiques.
.
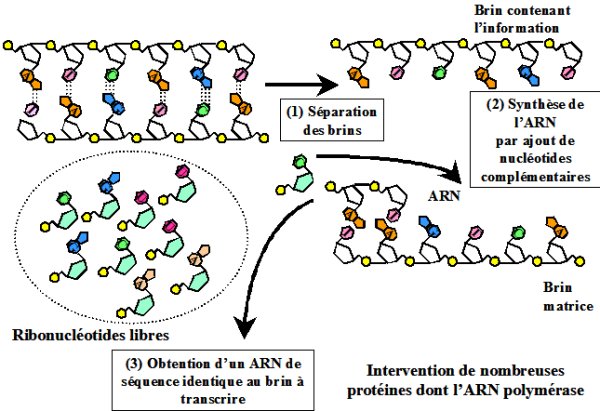
.
C) Traduction de l’ARN intermédiaire en protéines
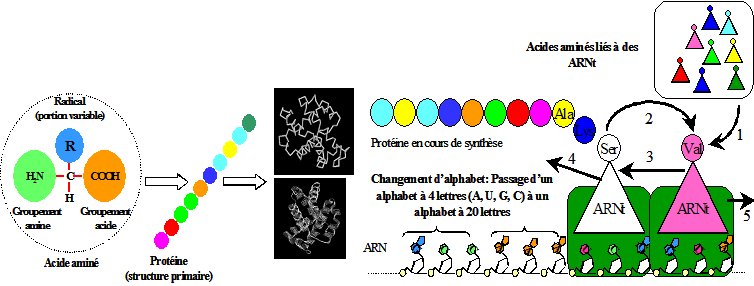
Seuls un petit nombre des ARN intermédiaires fabriqués à partir de l’ADN sont conservés sous forme d’ARN. La majorité d’entre eux sont traduits en protéines. Les protéines sont des polymères non ramifiés d’acides aminés remplissant des fonctions fondamentales dans la cellule (catalyseur de réactions chimiques, maintient des structures, génération et détection de mouvements, processus informationnels : réplication, transcription, traduction, régulation de l’expression des gènes…). Il existe 20 types d’acides aminés. Chaque acide aminé est construit sur le même schéma : un atome de carbone central sur lequel se greffent un groupement acide (COOH), un groupement amine (NH2), un radical et un atome d’hydrogène. C’est la nature du radical qui spécifie le type d’acide aminé. Chaque protéine possède une séquence linéaire (séquence primaire) d’acides aminés propre qui va dépendre de la séquence de l’ARN à partir de laquelle elle a été fabriquée. Cette chaîne linéaire va se replier adoptant une structure 3D qui donne à la protéine ses propriétés (Figure 4). Lors de la traduction, les nucléotides portés par l’ARN intermédiaire vont être « lus » par un complexe macromoléculaire : le ribosome. Ce système est composé d’ARN et de nombreuses protéines. Le ribosome lit les nucléotides 3 par 3 (on parle de triplet de nucléotides ou de codon) de manière non chevauchante. Les ARNt, petites molécules d’ARN, sont liés à un acide aminé spécifique à une extrémité, et possèdent à l’autre extrémité une séquence particulière de 3 nucléotides appelée anticodon. Cet anticodon va essayer de se lier au codon examiné par le ribosome. Si l’anticodon de l’ARNt est complémentaire du codon considéré, alors l’acide aminé qu’il porte va être incorporé dans la chaîne en cours de synthèse (Figure 5).
D) Régulation de l’expression de l’information génétique
Les molécules d’ADN sont de grande taille et contiennent des milliers de gènes (chacun spécifiant une protéine ou un ARN). Cependant, la cellule n’exprime pas constamment l’ensemble de ses gènes. Elle ajuste le taux de transcription et de traduction des différents gènes en fonction de ses besoins et des conditions du milieu grâce à un système de régulation très perfectionné. Ce système de régulation consiste en une grande diversité de protéines régulatrices capables de se lier à des régions non codantes qui parsèment l’ADN du génome. La liaison protéine régulatrice – séquence régulatrice va permettre l’expression du ou des gènes associés ou contraire l’inhiber. La quantité et l’organisation des séquences régulatrices varie d’un organisme à l’autre mais la stratégie de base et la même. Le génome cellulaire ne dicte pas seulement la nature des protéines que la cellule est capable de fabriquer mais aussi le moment et l’endroit où elles doivent être fabriquées.
E) Besoin d’énergie libre + matériaux bruts
Une cellule est un système éloigné de l’équilibre chimique : elle possède une grande quantité énergie interne. Cela signifie que si on la laisse mourir et se dégrader jusqu’à atteindre l’équilibre une grande quantité d’énergie sera libérée. L’approvisionnement en énergie est donc indispensable pour que la cellule puisse se maintenir (synthèse d’ADN, ARN de protéines, de lipides, division cellulaire, mouvement…). En plus d’une source d’énergie, elle a besoin de matériaux bruts à partir desquels elle fabriquera ses propres constituants. Même si toutes les cellules utilisent les mêmes constituants cellulaires de base (sucres, lipides, ADN, protéines, nucléotides, acides aminés…) et le même transporteur d’énergie (la molécule d’ATP), les sources d’énergie ainsi que les matériaux bruts importés diffèrent selon les cellules. Par exemple, certaines cellules comme les cellules végétales utilisent l’énergie solaire et quelques nutriments très simples pour fabriquer l’ensemble de leurs constituants alors que d’autres comme les animaux, qui se nourrissent d’autres êtres vivants, dont ils tirent l’énergie et une grande partie des molécules organiques qu’ils sont incapables de fabriquer (on parle d’auxotrophie). D’autres cas plus extrêmes concernent certains pathogènes qui ont perdus la capacité de fabriquer un grand nombre (voir la quasi totalité) des molécules de base de leurs constituants. Ils prélèvent les molécules dont ils ont besoins auprès de leur hôte (voir le cas des mycoplasmes ci-dessous).
F) Délimitation spatiale par une membrane plasmique
Toutes les cellules sont entourées d’une membrane plasmique semi-perméable qui maintient son intégrité et qui agit comme une barrière sélective qui permet à la cellule d’importer des matériaux bruts, de concentrer ses produits de fabrication et d’exporter ses déchets. Elle est constituée de principalement de lipides et de protéines spécialisées qui permettent le transport spécifique de molécules au travers de la membrane. Le type des protéines membranaires va déterminer quel type de molécules la cellule est capable d’importer.
II. La cellule minimale
1/ Définition du problème
La question de la définition d’une cellule minimale peut se résumer ainsi : « Quel est le plus petit set de gènes nécessaires et suffisants pour maintenir une forme de vie cellulaire moderne, c’est-à-dire capables d’assurer toutes les fonctions cellulaires décrites ci-dessus et possédant les 3 caractéristiques du vivant ». Les tentatives de réponses se sont limitées à la définition du protéome minimal (c’est à dire à l’ensemble des protéines nécessaires à la cellule minimale) en ne tenant pas compte des systèmes de régulations et des régions non codantes existant dans les chromosomes dont toute la complexité n’est pas encore comprise. Implicitement on suppose que cette cellule minimum est placée dans les conditions les plus favorables (1) en présence de tous les nutriments essentiels dont elle pourrait avoir besoin (i.e. cette cellule n’a donc pas besoin de les fabriquer) et (2) en absence de tout stress environnemental. Ceci implique qu’elle n’est pas censée survivre à tous les changements de l’environnement auxquels serait confrontée une cellule réelle. Intuitivement on s’attend à ce que ce set gènes recouvre l’ensemble des grandes fonctions communes à toutes les cellules sans pour autant savoir combien de gènes sont nécessaires au maintien de chaque fonction. La recherche de la cellule minimum a commencé à la fin des années 60. Depuis deux grands axes de recherche ont été développés pour appréhender cette question : (1) les méthodes expérimentales (in vivo) et (2) les méthodes de bioinformatique de comparaison du contenu en gènes des génomes cellulaires (in silico).
A) Méthodes expérimentales
a. Principe :
Identifier les gènes indispensables dans des conditions de croissance particulières chez des organismes modèles en générant des mutations classées en fonction de leur létalité : Un gène qui lorsqu’il est muté donne un phénotype létal sera qualifié d’indispensable et de non-indispensable sinon.
.
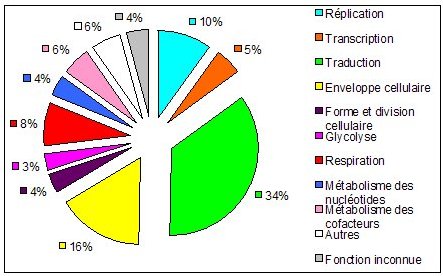
* Stratégies de mutagenèse massives par des transposons : L’objectif est de saturer le génome des cellules par l’insertion de transposons (séquences d’ADN) qui vont inactiver les gènes lorsqu’elles s’insèrent à l’intérieur de ces derniers. Si des cellules sont capables de croître malgré tout, cela signifie qu’aucun transposon n’a atteint de gènes indispensables. Les gènes pour lesquels aucune insertion n’est observable sont identifiés comme indispensables. Ce type d’étude a été réalisé chez Mycoplasma genitalium, une bactérie pathogène à faible taux de G+C qui possède l’un des plus petits génomes cellulaires connus (480 gènes pour une taille de 540kb). A cause de son très petit génome il était supposé que le génome de M. genitalium est composé principalement de gènes indispensables (par exemple 1/3 de ses gènes interviennent dans la fabrication des protéines, Figure 5). De manière surprenante, cette étude montre que 180-215 gènes présents chez M. genitalium seraient non-indispensables ce qui représente 1/3 à 1/2 de ses gènes. Cependant comme attendu, les catégories fonctionnelles contenant la plus forte proportion de gènes indispensables sont celles liées aux processus cellulaires fondamentaux (fabrication des protéines, métabolisme des lipides, production d’énergie, reproduction, transport…). Il est intéressant de noter que 67 gènes indispensables ont des fonctions inconnues, impliquant que (1) tous les processus cellulaires fondamentaux n’ont pas encore été identifiés et/ou (2) que ces gènes correspondent à des fonctions connues mais n’y ont pas été reliés. Une interprétation plus fine ces résultats nécessiterait un travail expérimental poussé, mais on peut déjà voir émerger l’idée que le set de gènes indispensables est dépendant de l’environnement dans lequel on le définit. Par exemple le fait que 60 % des gènes impliqués dans l’enveloppe cellulaire soient non-indispensables peut signifier qu’ils peuvent être importants pour la cellule dans son environnement naturel (lors de l’infection de son hôte) mais inutiles dans les conditions de culture en laboratoire.
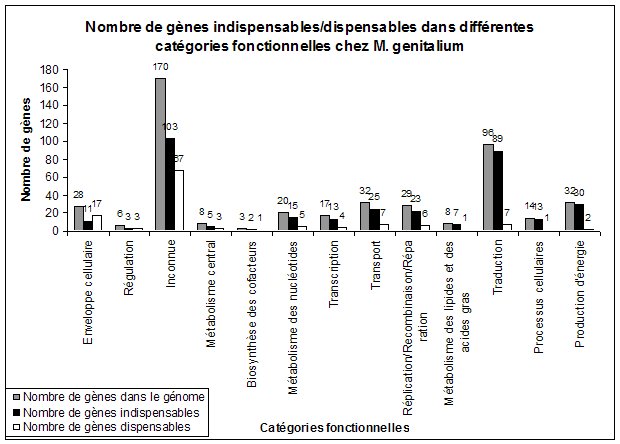
.
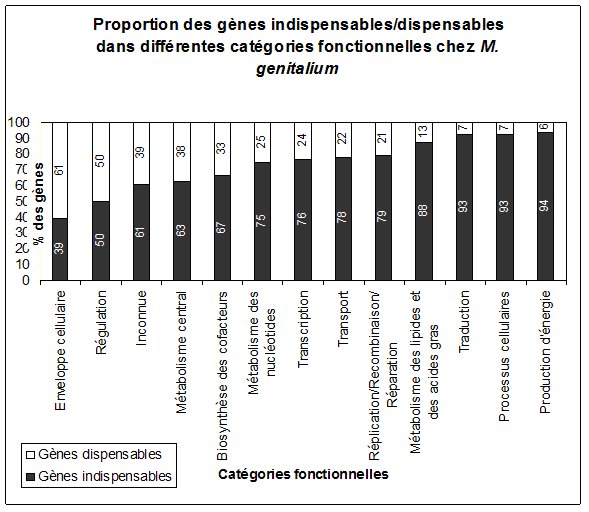
.
* L’inhibition de l’expression des gènes par l’utilisation d’ARN anti-sens : Principe : Stratégie consistant à bloquer la traduction des ARN intermédiaires sans que la séquence du gène correspondant ne soit altérée. Elle est basée sur la fabrication d’ARN dits « anti-sens » complémentaires de la séquence d’un ARN produit par le gène étudié. Lorsque de dernier est transcrit, l’ARN produit va s’associer avec l’ARN ani-sens provoquant un blocage de la traduction.
Une étude a été menée chez Staphylococcus aureus (une bactérie pathogène gram positive à bas taux de G+C comptant 2595 gènes) 7. Parmi les milliers d’ARN anti-sens produits, ceux correspondants à 658 gènes induisent un phénotype l’étal. La comparaison de ces 658 gènes potentiellement indispensables avec les gènes présents chez M. genitalium montre que 168 ont été identifiés comme indispensables chez ce dernier (cf. mutagenèse par transposons) et se répartissent dans un petit nombre de catégories fonctionnelles (Tableau 2, Figure 7 et 8). Il peut paraître surprenant que la majorité des gènes indispensables chez S. aureus (658-168=490) soit absents chez M. genitalium. Cela montre qu’il existe des différences majeures entre les organismes : des gènes indispensables chez l’un ne le seront peut-être pas chez l’autre. Par exemple M. genitalium ne possède pas paroi contrairement à S. aureus. Il apparaît donc normal que des gènes indispensables au maintient de la paroi chez S. aureus soient absent chez M. genitalium.
.
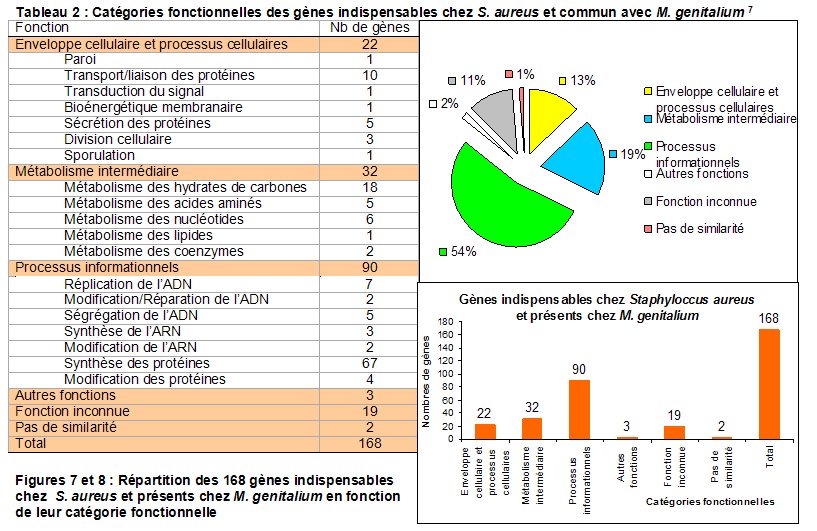
.
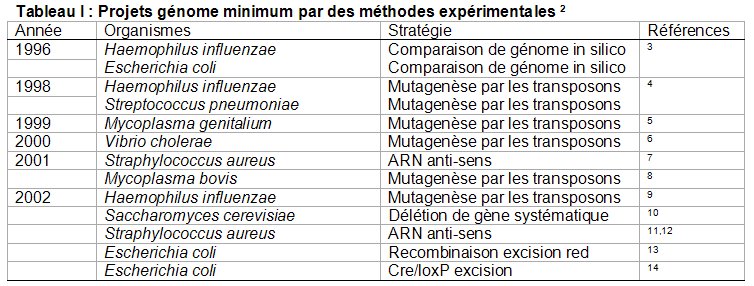
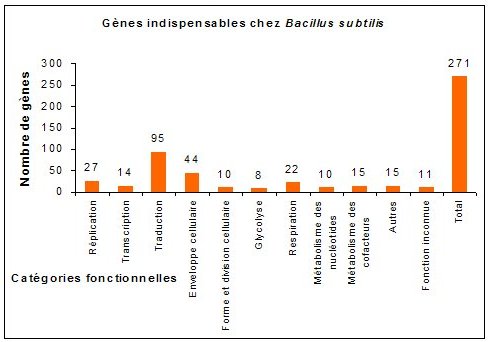
* L’inactivation systématique des gènes présents dans un génome : Cette méthode consiste à remplacer la séquence d’un gène dans un génome par une séquence inactive de ce même gène. Elle a été appliquée chez Bacillus subtilis (organisme modèle bactérien appartenant au groupe des bactéries gram positives à bas taux de G+C, 15). Le génome de cette bactérie contient 4100 gènes parmi lesquels 271 seraient indispensables pour la croissance de B. subtilis lorsqu’ils sont inactivés un par un. 80% de ces gènes essentiels appartiennent à quelques grandes catégories fonctionnelles clés (Tableau 3, Figures 9 et 10).
b. Limites de ces approches :
Même si ces méthodes expérimentales donnent des résultats intéressants, elles possèdent un certain nombre de limites. Par exemple, l’insertion des transposons sous-estime très probablement la quantité de gènes réellement essentiels car certains d’entre eux peuvent être résistants à l’insertion de transposons et être classés à tort dans la catégorie des gènes non-essentiels. L’utilisation de d’ARN anti-sens peut conduire à une surestimation du nombre de gènes indispensables dans le cas d’ARN polycistroniques (ARN intermédiaire unique permettant la traduction simultanée de plusieurs protéines). L’ARN anti-sens d’un gène non essentiel situé dans un ARN polycistronique peut conduire à la non-traduction de l’ensemble des protéines (dont certaines peuvent être essentielles). De plus un ARN anti-sens complémentaire d’un motif partagé par un ensemble de gènes peut inhiber la totalité des gènes partageant ce motif. L’inactivation de gènes chez B. subtilis qui possède un gros génome peut biaiser l’estimation du nombre de gènes indispensables, car il a été montré que une fonction essentielle peut parfois être assurée par plusieurs gènes présents dans le génome (c’est ce qu’on appelle la redondance génomique). Ce phénomène est très fréquent dans le cas de gènes paralogues (gènes issus d’un événement de duplication et pouvant avoir conservé des fonctions proches), or 50% du génome de B. subtilis est composé de gènes paralogues. Enfin il ne faut pas oublier qu’elles permettent de définir le set de gènes essentiels à un organisme bactérien précis dans des conditions environnementales fixées. D’un environnement à l’autre et d’un organisme à l’autre, la composition de ce set minimum de gènes peut varier. De même, le fait qu’un gène soit indispensable ou non dépend du contexte génomique dans lequel il se trouve. Il existe des inactivations mutuellement exclusives. Par exemple la délétion du gène X ou du gène Y peut être tolérée individuellement suggérant que ni X ni Y n’est indispensable mais pas la délétion simultanée de X et Y qui induit un phénotype létal. Par exemple la délétion des deux gènes nrdA et nrdD chez E. coli inhibe sa croissance en aérobie et en anaérobie conduisant à un phénotype létal. Ceci illustre que le set minimal de gènes indispensables n’est pas équivalent au génome minimum car des gènes non essentiels individuellement ne le sont pas collectivement. Inversement il peut exister des délétions inclusives, l’inactivation du gène X ou du gène Y conduit à un phénotype létal suggérant que ces gènes sont indispensables alors que leur délétion simultanée peut conduire à un phénotype viable. Par exemple il a été mis en évidence chez E. coli un système toxine / antitoxine. La délétion du gène codant pour l’anti-toxine conduit à la mort des cellules, alors que la délétion combinée du gène codant pour la toxine et l’anti-toxine restore la viabilité. Pour réaliser des études vraiment fines il faudrait étudier l’inactivation de combinaisons de gènes et non de gènes individuels ce qui est impossible actuellement.
c. Discussion :
Les approches expérimentales ont été réalisées principalement sur des cellules bactériennes car elles possèdent déjà des petits génomes. En dépit de leurs limitations, une cellule minimale semble en émerger : c’est une cellule bactérienne simple consistant en un compartiment délimité par une membrane et réalisant les fonctions caractéristiques des cellules : (1) la réplication, (2) la division du compartiment, (3) la fabrication de protéines, et (4) un métabolisme sommaire pour la production d’énergie (la glycolyse). Il est important de noter la totale absence de facteurs de régulation de l’expression des gènes. Le nombre de gènes estimés varie de 168 à 500. 80% des gènes essentiels chez B. subtilis sont présents chez toutes les bactéries ayant des génomes d’une taille supérieure à 3 Mb alors que seuls 57 % sont trouvés chez toutes les bactéries. Les gènes qui sont absents des génomes bactériens de petite taille sont ceux impliqués dans la synthèse de la paroi cellulaire, la forme et la division cellulaire, le métabolisme et la fabrication de nombreux composants. Ceci n’est pas surprenant car les plus petits génomes bactériens correspondent à des pathogènes au mode de vie ultra-spécialisé, qui ont perdu un grand nombre de gènes et se retrouvent avec de nombreuses auxotrophies. Il résulte également de ces études que les gènes indispensables sont très souvent présents en un seul exemplaire dans les génomes alors que ceux qui présentent le plus fort taux de redondance sont souvent non indispensables. Ceci peut paraître paradoxal car on pourrait s’attendre à ce que les gènes impliqués dans des fonctions cellulaires indispensables soient fortement redondants (cela permet de limiter les dégâts en cas de perte de l’un d’entre eux). Ceci suggère que la stratégie adoptée par ces organismes concernant la redondance des gènes qui est plus axées sur l’adaptation à des changements de conditions environnementales que sur la « sauvegarde » des gènes assurant des fonctions cellulaires indispensables.
B) Comparaison du contenu en gènes des génomes
a. Principe : Cette approche repose sur l’idée simple et astucieuse que toutes les cellules actuelles possèdent une caractéristique commune : ce sont des cellules. Cette caractéristique a été héritée d’un même ancêtre commun, le dernier ancêtre commun à toutes les cellules actuelles qu’on appelle LUCA. Il semble donc logique de supposer que toutes les cellules ont également héritées de LUCA de l’ensemble des gènes nécessaires à la fabrication et au fonctionnement d’une cellule (ces derniers ainsi que de nombreux gènes accessoires variables d’une cellule à l’autre constituent les génomes modernes). Donc en comparant le contenu en gènes des génomes modernes, il est théoriquement possible d’identifier tous les gènes hérités de ce dernier ancêtre commun et qui ont été retenus par toutes les cellules au cours de l’évolution. Compte tenu de la grande capacité des procaryotes à perdre des gènes au cours de l’évolution, il est très probable que ces gènes conservés chez tous les êtres vivants sont majoritairement ceux qui définissent l’essence de la cellule. Cependant les choses ne sont pas aussi simples car au cours de l’évolution peuvent se produire ce qu’on appelle des remplacements non-orthologues de gènes. Ceci a pour conséquence que des gènes non-orthologues peuvent assurer des fonctions similaires au sein des cellules 17, compliquant l’identification de ce qui est conservé entre les génomes.
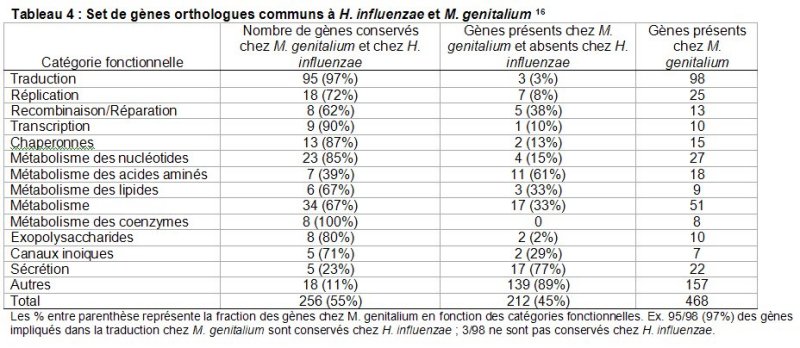
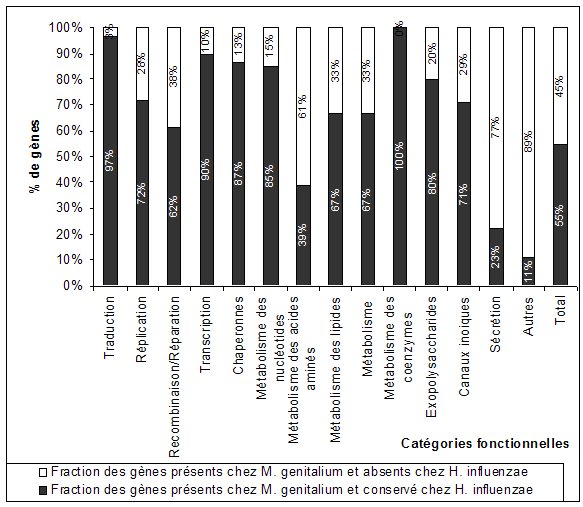
La première mise en œuvre date de 1996 et recherchait les orthologues conservés chez deux bactéries pathogènes M. genitalium et d’Haemophilus influenzae (468 gènes pour 0.58 Mb et 1700 gènes pour 1.83 Mb, respectivement) qui ont divergé à partir d’un ancêtre commun il y a plus de 1.5 milliard d’années 16. Le protocole suivit est le suivant : (1) identifier tous les orthologues communs aux deux génomes (233 gènes), (2) étudier les « trous » dans les fonctions cellulaires clés afin d’identifier les remplacements non orthologues potentiels et compléter la liste des gènes (23 gènes) et (3) éliminer les gènes apparemment redondants et/ou spécifiques du mode de vie parasite (6 gènes).
.
Les % entre parenthèse représente la fraction des gènes chez M. genitalium en fonction des catégories fonctionnelles. Ex. 95/98 (97%) des gènes impliqués dans la traduction chez M. genitalium sont conservés chez H. influenzae ; 3/98 ne sont pas conservés chez H. influenzae.
b. Discussion :
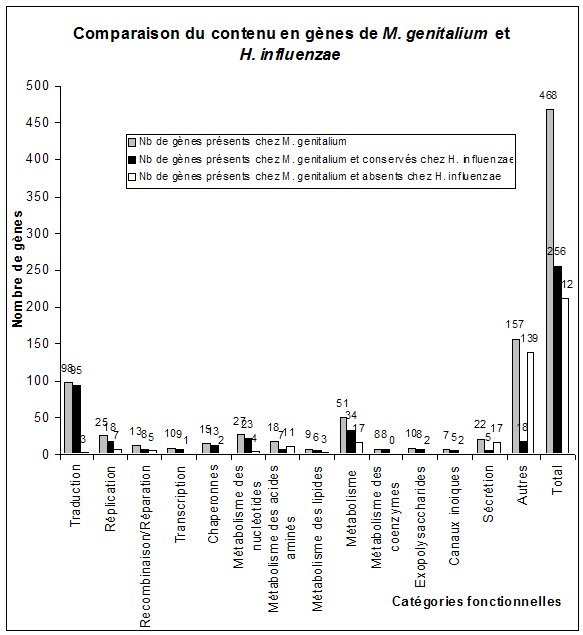
Cette approche a permis d’identifier dans ces deux bactéries 256 gènes conservés supposés être nécessaires et suffisants pour permettre une vie cellulaire moderne et être proche du set minimum de gènes. Il est important de noter que ce chiffre est assez compatible avec ceux obtenus par les méthodes expérimentales. En particulier la cellule théorique contient (1) un système presque complet de traduction ; (2) une machinerie de réplication de l’ADN ; (3) un système rudimentaire de réparation et de recombinaison de l’ADN ; (4) un système de transcription presque complet mais sans de facteur de régulation ; (5) des chaperonnes ; (6) un métabolisme intermédiaire anaérobie virtuellement restreint à la glycolyse ; (7) pas de système de synthèse des acides aminés ; (8) pas de système de synthèse de novo des nucléotides ; (9) une voie de biosynthèse des lipides limitée (absence de synthèse des acides gras) ; (10) un système d’exportation des protéines et (11) un répertoire limité de protéine de transport de métabolites (Tableau 4 et Figures 11 et 12). Les besoins nutritifs d’une telle cellule sont assez considérables si on considère qu’elle aurait besoin d’importer tous ses acides aminés, tous ses nucléotides, tous ses acides gras et ses coenzymes complexes.
Figures 10 et 11 : Nombre / Fraction des gènes conservés chez M. genitalium et conservés ou absents chez H. influenzae dans les différentes catégories fonctionnelles
.
c. Limites de cette approche :
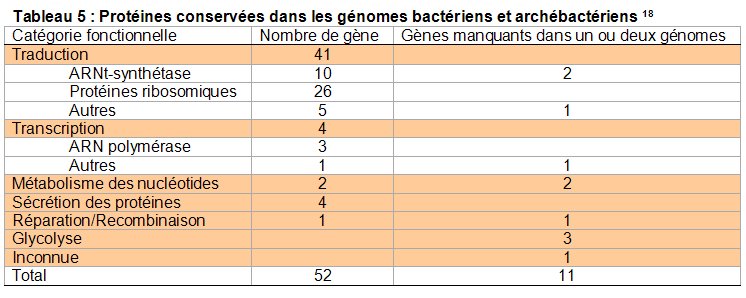
Ce qui a été reconstruit par cette approche est probablement plus un génome bactérien minimum (puisquecetteanalysecomparedeux génomes bactériens) qu’un génome minimal cellulaire en général car il est très probable quece génome minimal comporte des solutionsbactériennespour certaines fonctions. De plus certains de ces gènes prédits comme indispensables ont pu être inactivésexpérimentalement par l’insertionde transposons suggérantquecertains de ces gènes ne sont pas aussi indispensables que ce qui avait été prédit. De plus, selon les organismes comparés le set de gènes orthologues peut varier de manière considérable. En particulier, l’inclusion d’un plus grand nombre de génomes très divergents dans ce style d’analyse diminue très fortement la taille du set de gènes conservés entre les génomes. Par exemple, la même étude appliqué aux sets de protéines codées par 21 génomes microbiens et archébactériens, aboutit à une liste de 52 gènes (Tableau 5). Cette liste de protéines comprend principalement des protéines impliquées dans la formation du ribosome et la biosynthèse des protéines, mais n’est pas suffisante pour construire une cellule cohérente ni même un ribosome fonctionnel. En effet, un biais important a été introduit dans cette étude : l’inclusion de plusieurs parasites obligatoires, qui ont une biochimie extrêmement simplifiée. Cependant si une tolérance de une à deux absences est incluse, on peut ajouter 11 gènes et du coup des fonctions cellulaires préalablement absentes, comme la glycolyse ou un métabolisme des nucléotides, commencent à apparaître.
Finalement, se pose également le problème de la trop grande divergence entre les orthologues très éloignés (au-delà de toute reconnaissance) ainsi que du remplacement non homologue de gènes (ce phénomène qui touche potentiellement toutes les classes d’enzymes et tous les processus biologiques pourrait atteindre 5% des gènes présents dans le génome minimal). C’est pourquoi une approche légèrement différente mais surtout moins restrictive a été proposée : la recherche de fonctions universelles conservées qui ne se focalise plus sur la recherche d’orthologues conservés entre les génomes mais sur l’identification de groupes d’orthologues (COG) impliqués dans des fonctions semblables. Cette approche a été permis par le développement d’une base de données de clusters d’orthologues associés à des annotations fonctionnelles 19. En examinant ces clusters on peut remarquer que 327 paires clusters « universels » ont des annotations fonctionnelles similaires. Par exemple les clusters de gènes orthologues COG0406 et COG0696 contiennent des phosphoglyceromutases et illustrent le cas probable d’un remplacement non-orthologue (alternatives évolutives pour la réalisation de la même fonction). Parallèlement à l’utilisation des COGs, ce sont les groupes d’organismes (et non plus organismes individuels) qui sont considérés ce qui limite considérablement l’impact des multiples auxotrophies des organismes parasites. La cellule inférée par la recherche de fonctions universelles conservées possèderait quelques 320 gènes et pourrait synthétiser de novo ses acides aminés, ses nucléotides, des carbohydrates complexes et quelques coenzymes et ne dépendre plus que d’un petit nombre de précurseurs prélevés dans l’environnement.
Les grandes catégories fonctionnelles absentes du génome minimal ne sont pas de peu d’importance. Par exemple, plusieurs protéines impliquées dans la réplication de l’ADN ne sont pas orthologues entre les bactéries d’une part et les eucaryotes/archébactéries d’autre part. Les explication proposées sont que (1) l’ADN et les protéines qui lui sont associées sont apparus indépendamment dans la lignée bactérienne et dans la lignée archées + eucaryotes, (2) que le dernier ancêtre commun possédait un génome à ADN avec un système de réplication très rudimentaire (on trouve quelques protéines clairement homologues entre les trois domaines), mais que dans ce cas là, la réplication a évolué dans différentes directions après la divergence archées + eucaryotes et bactéries par recrutement indépendant de protéines ou (3) que des remplacements non orthologues à partir d’un ancêtre commun à ADN se sont produits 20. De même peu d’enzymes de réparation de l’ADN sont orthologues entre les trois domaines et peu d’enzymes impliquées dans la biosynthèse des lipides sont orthologues entre eucaryotes/bactéries et archées (ce qui est en accord avec l’observation de différences entre les lipides composants la membrane de ces organismes). Quels que soient les mécanismes impliqués et la lignée dans laquelle le remplacement non orthologue a eu lieu, le génome d’une cellule minimale doit remplir l’ensemble des fonctions caractéristiques de la cellule et doit donc être complété par des gènes pouvant les assurer. L’inclusion des solutions eucaryotes, bactériennes ou archébactériennes pour certaines fonctions conduira à des alternatives pour la construction du génome minimum.
.
III. Peut-on définir le génome minimum ?
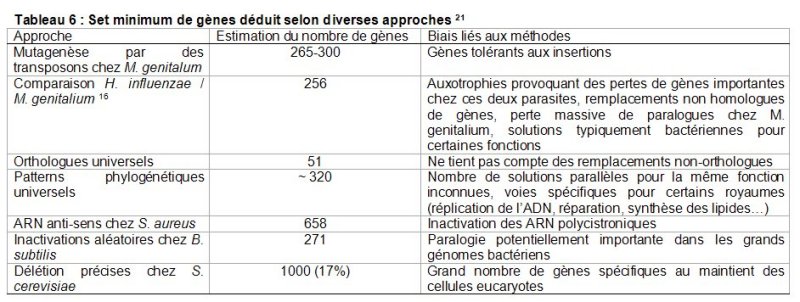
Les différentes approches développées pour définir une cellule minimale sont relativement congruentes du point de vue du contenu moyen en gènes (Tableau 6). Un consensus autour de 250-300 gènes semble raisonnable.
Il est intéressant de noter que plusieurs fois au cours de l’évolution, des phénomènes de réduction génomique se sont produits indépendamment aboutissant à des génomes très réduits et différents selon les lignées considérées. La perte de gènes peut être vue d’un point de vue évolutif comme l’engagement dans des voies à sens unique : la perte d’un ou de plusieurs gènes peut entraîner des changements irréversibles que même l’acquisition de nouveaux gènes ne saurait compenser. Par exemple les mycoplasmes ont perdu de nombreux gènes impliqués dans la régulation de la transcription. Ceci a peut-être été rendu possible par un déplacement de la régulation de l’expression des gènes au niveau de la traduction. De même la perte d’un très grand nombre de transporteurs chez M. genitalium a peut-être accru la pression de sélection pour la conservation des transporteurs ayant une large spécificité de substrat.. De même, la perte de gènes peut limiter le nombre et le type de pertes qui seront tolérées dans le futur, c’est pourquoi la définition d’un génome minimum à partir de l’expérimentation sur matériel vivant va donc aboutir à des résultats différents selon les organismes considérés, car selon les lignées, ce n’est pas le même répertoire de gènes qui est présent. De même, la relation étroite entre environnement et génome va influer sur la réponse en définissant ce qui est indispensable et ce qui ne l’est pas, relativement à un environnement : selon l’environnement différentes solutions au problème du génome minimum pourront être proposées.
Malgré une réduction génomique parfois très importante, les plus petits génomes actuels comme ceux des mycoplasmes restent beaucoup plus grands que les génomes minimaux prédits par des méthodes bioinformatiques (même si on retire les 10% de gènes spécifiques liés à leur mode de vie parasitaire). La différence qu’il reste entre ces génomes réduit et les génomes minimaux inférés reflète probablement la différence qu’il existe entre les génomes résultants de l’interaction de gènes pendant des millions d’années d’évolution et ayant subit une histoire évolutive et un génome qui serait fabriqué de novo par un ingénieur en rassemblant artificiellement des choses qui ont été inventées indépendamment dans différentes lignées évolutives (mélange de solutions eucaryotes, bactériennes et archébactériennes) pouvant même jusqu’à inventer de nouvelles solutions de toutes pièces pour contourner un problème donné.
Quoiqu’il en soit, il ressort des nombreuses études réalisées, qu’il est très probable qu’en pratique nous ne pourrons jamais proposer une réponse unique au problème de la cellule minimum car c’est en réalité une construction purement théorique, une abstraction. Ce n’est pas quelque chose qui a été expérimenté par la nature car elle requiert un environnement idéal libre de toute pression de sélection et pourvoyant à tous ses besoins. En pratique il existe un grand nombre de solutions possibles au problème de la cellule minimum qui dépendent du type de cellulaire considéré : on peut définir un génome minimum pour chaque type de métabolisme : quel est le génome minimal pour une cellule phototrophe capable de croître uniquement en présence de CO2, de lumière et sels inorganiques, quel est le génome minimum pour une cellule hétérotrophe simple (capable de croître uniquement en présence d’un milieu de base contenant une seule source de carbone organique comme le glucose et des sels inorganiques, quel est le génome minimal d’une cellule hétérotrophe plus complexe nécessitant des milieux de culture plus riches (parfois même incomplètement caractérisés, comme du sérum), quel est le génome minimum d’un parasite cellulaire obligatoire ?…
.
IV Bibliographie
- 1 Luisi, P.L. (1998) About various definitions of life. Origin of Life and Evolution of the Biosphere 28, 613-622
- 2 Smalley, D.J. et al. (2003) In search of the minimal Escherichia coli genome. Trends Microbiol 11 (1), 6-8
- 3 Lipton, M.S. et al. (2002) Global analysis of the Deinococcus radiodurans proteome by using accurate mass tags. Proc Natl Acad Sci U S A 99 (17), 11049-11054
- 4 Sassetti, C.M. et al. (2001) Comprehensive identification of conditionally essential genes in mycobacteria. Proc Natl Acad Sci U S A 98 (22), 12712-12717
- 5 Giaever, G. et al. (2002) Functional profiling of the Saccharomyces cerevisiae genome. Nature 418 (6896), 387-391
- 6 Akerley, B.J. et al. (2002) A genome-scale analysis for identification of genes required for growth or survival of Haemophilus influenzae. Proc Natl Acad Sci U S A 99 (2), 966-971
- 7 Forsyth, R.A. et al. (2002) A genome-wide strategy for the identification of essential genes in Staphylococcus aureus. Mol Microbiol 43 (6), 1387-1400
- 8 Hutchison, C.A. et al. (1999) Global transposon mutagenesis and a minimal Mycoplasma genome. Science 286 (5447), 2165-2169
- 9 Akerley, B.J. et al. (1998) Systematic identification of essential genes by in vitro mariner mutagenesis. Proc Natl Acad Sci U S A 95 (15), 8927-8932
- 10 Bochner, B.R. et al. (2001) Phenotype microarrays for high-throughput phenotypic testing and assay of gene function. Genome Res 11 (7), 1246-1255
- 1 Judson, N. and Mekalanos, J.J. (2000) TnAraOut, a transposon-based approach to identify and characterize essential bacterial genes. Nat Biotechnol 18 (7), 740-745
- 12 Judson, N. and Mekalanos, J.J. (2000) Transposon-based approaches to identify essential bacterial genes. Trends Microbiol 8 (11), 521-526
- 13 Kolisnychenko, V. et al. (2002) Engineering a reduced Escherichia coli genome. Genome Res 12 (4), 640-647
- 14 Yu, B.J. et al. (2002) Minimization of the Escherichia coli genome using a Tn5-targeted Cre/loxP excision system. Nat Biotechnol 20 (10), 1018-1023
- 15 Kobayashi, K. et al. (2003) Essential Bacillus subtilis genes. Proc Natl Acad Sci U S A 100 (8), 4678-4683
- 16 Mushegian, A.R. and Koonin, E.V. (1996) A minimal gene set for cellular life derived by comparison of complete bacterial genomes. Proc Natl Acad Sci U S A 93 (19), 10268-10273
- 17 Galperin, M.Y. et al. (1998) Analogous enzymes : independent inventions in enzyme evolution. Genome Res 8 (8), 779-790
- 18 Huynen, M.A. and Bork, P. (1998) Measuring genome evolution. Proc Natl Acad Sci U S A 95 (11), 5849-5856
- 19 Tatusov, R.L. et al. (1997) A genomic perspective on protein families. Science 278 (5338), 631-637
- 20 Forterre, P. (1999) Displacement of cellular proteins by functional analogues from plasmids or viruses could explain puzzling phylogenies of many DNA informational proteins. Mol Microbiol 33 (3), 457-465
- 21 Mushegian, A. (1999) The minimal genome concept. Curr Opin Genet Dev 9 (6), 709-714
_________________________________________
L’auteur, Céline Brochier, est maître de conférences à l’université de Provence Aix-Marseille 1, en biologie. Elle est spéacialiste de la phylogénie des êtres vivants.
.





Bonjour,
Je me permets de vous contacter parce que je me posais une question – à propos de la dynamique du vivant – à partir d’une histoire qui n’avait rien à voir avec la biologie.
Je n’ai pas de formation scientifique – d’où ce courrier un peu informel.
Dans le cadre d’une recherche sur l’invention de la photographie – la question était la suivante : que faut-il pour faire un enregistrement ?
On peut très simplement décrire un système d’enregistrement de cette manière : il faut (au moins) un capteur – un circuit d’inscription et un support – il faut l’agencement de ces trois éléments. Pour une photographie – le capteur (de type chimique – l’émulsion) – un circuit d’inscription (la chambre noire) et un support (papier – plaque de verre, etc…) Bien sûr les capteurs – les circuits d’inscription et les supports peuvent changer ou varier mais le schéma de base reste le même quelque soit la » simplicité » ou la » complexité » du système.
Il faut souligner le fait qu’un système d’enregistrement permet souvent de répliquer l’enregistrement lui même – qu’il y a si l’on veut un prolongement direct entre la possibilité d’enregistrer et de répliquer – en l’occurrence une image. Et aussi qu’à partir d’un enregistrement donné les modes de réplication peuvent changer.
Je me disais qu’il n’y avait peut-être aucune raison – s’il on s’en tient à ce schéma – de considérer l’enregistrement comme une invention exclusivement technique – puisque les systèmes biologiques semblent avoir tout ce qu’il faut pour fonctionner comme de bons vieux systèmes d’enregistrement reproductibles.
Cette analogie m’a étonné – je me demande si l’on peut considérer certaines transformations au cours de l’évolution en rapport avec des changements intervenus dans les trois pôles de cet agencement – supports – circuit d’inscriptions et capteurs ?
Peut-on voir une cellule comme cela – avec ses capteurs – ses circuits d’inscription et ses supports ? Bien sûr cette description globale n’explique rien des processus mais cela élargirait mon champ de réflexion. J’aimerais donc savoir si l’on peut dire cela – si un(e) biologiste peut souscrire à cette description globale…
Cordialement,
P F